
Diese Zusammenfassung beschreibt eine Studie, die sich darauf konzentriert, wie Graphen und Few-Layer-Graphen (FLG) mit Zellmembranen interagieren. Dies ist entscheidend für die Weiterentwicklung biomedizinischer Anwendungen und die Gewährleistung der Sicherheit im Umgang mit Graphen.
Die Forschung kombiniert experimentelle Techniken und Simulationen, um das Verhalten von Graphenmaterialien in biologischen Umgebungen zu untersuchen. Wichtige Ergebnisse und Methoden umfassen:
1. Bildgebung und Beobachtungen:
- Experimente mit konfokaler Fluoreszenz- und Elektronenmikroskopie zeigten, dass Graphen kante-zuerst in Zellen eindringt, was zu einer vollständigen Internalisierung führt.
- Dieses Verhalten wurde bei verschiedenen FLG-Proben mit lateralen Abmessungen von 0,5 bis 10 μm beobachtet.
2. Simulationen und Energiebarrieren:
- Molekulardynamik-Simulationen (sowohl grobkörnige, als auch auf Atomniveau) zeigten hohe Energiebarrieren für das direkte Eindringen von Graphenschichten in Membranen, was darauf hindeutet, dass ein einfacher Membrandurchtritt energetisch ungünstig ist.
3. Eindringmechanismus:
- Detaillierte Simulationen identifizierten, dass unregelmäßige Kanten, Ecken und Vorsprünge auf Graphenschichten lokal die Membran durchstechen. Diese scharfen Strukturen initiieren einen Prozess, bei dem sich die Membran entlang der Kante der Graphenschicht ausbreitet, wodurch die ansonsten hohe Energiebarriere umgangen wird.
4. Konsistenz der Zellaufnahme:
- Der vorgeschlagene Mechanismus erklärt, wie Zellen große Graphenschichten aufnehmen können, und stimmt mit den Bioimaging-Ergebnissen überein, die bei menschlichen Keratinozyten, Lungenepithelzellen und murinen Makrophagen beobachtet wurden.
Implikationen
Diese Studie liefert Erkenntnisse darüber, wie die physikalische Struktur von Graphen seine Interaktionen mit biologischen Membranen beeinflusst, und hebt die Bedeutung der Kantenstruktur für die Zellaufnahme hervor. Diese Ergebnisse sind relevant für die Entwicklung graphenbasierter biomedizinischer Werkzeuge und für die Bewertung potenzieller Gesundheitsrisiken im Zusammenhang mit Graphenexposition. (Gesundheit: 7 Geheimnisse über Krebs, die Sie nicht erfahren sollen)
Graphen ist ein 2D-plattenförmiges Material, das aus einer einzigen Schicht hexagonal angeordneter Kohlenstoffatome besteht und außergewöhnliche elektrische, mechanische und thermische Eigenschaften aufweist. Bilayer-, Trilayer-, Few-Layer-Graphen- und Mehrschichtstrukturen mit einer Dicke von <100 nm sind eng verwandte Nanomaterialien, die häufig durch thermische Exfoliation von Graphit hergestellt werden und während der Verarbeitung oft als Trockenschäume vorliegen, was die Wahrscheinlichkeit einer Exposition am Arbeitsplatz erhöht. In diesem Artikel beziehen wir uns auf diese Materialgruppe als „Graphenfamilien-Nanomaterialien“ (GFNs).
Viele GFNs haben aerodynamische Durchmesser von weniger als 5 μm und fallen daher in die Kategorie potenziell einatembarer Materialien. Selbst GFNs mit großen lateralen Abmessungen (>20 μm) liegen typischerweise im potenziell einatembaren Bereich, und bei diesen stellen die großen lateralen Abmessungen Herausforderungen für die makrophagenvermittelte Clearance dar, was auf lange Verweilzeiten in der Lunge hindeutet.
Messungen am Arbeitsplatz und Laborsimulationen haben das Potenzial für die Exposition des Menschen gegenüber synthetischen Nanomaterialien durch Inhalation während mehrerer Schritte im Herstellungsprozess dokumentiert. Neben der Inhalation von Trockenschäumen können kohlenstoffbasierte Nanomaterialien auch während der Sonikation von Partikeln in Suspension, sowie beim Schneiden oder Bohren von Verbundwerkstoffen in die Luft gelangen. Neben beruflichen Expositionen können GFNs gezielt implantiert oder injiziert werden, beispielsweise für biomedizinische Anwendungen wie Biosensoren, Gewebegerüste, Träger für die Arzneimittelabgabe oder Gentherapie, antibakterielle Wirkstoffe und Bioimaging-Sonden. Die große spezifische Oberfläche von Graphen ermöglicht eine hochdichte Biofunktionalisierung oder Wirkstoffbeladung, und eine effizientere Tumorzielgenauigkeit. Zudem kann Graphen eine geringere Toxizität und bessere Herstellungsreproduzierbarkeit als einige andere Materialplattformen bieten.
Die lateralen Abmessungen von GFNs erstrecken sich über mehrere Größenordnungen, von 10 nm bis >100 μm (größer als die meisten Zielzellen). Die maximalen Abmessungen eines Nanomaterials sind entscheidend für die Zellaufnahme, die renale Clearance, den Transport über die Blut-Hirn-Schranke und viele andere biologische Phänomene. Graphenbasierte Schichten mit kleinen lateralen Abmessungen (<100 nm) werden für Arzneimittelabgabe- und Diagnoseanwendungen entwickelt. Es wurde beobachtet, dass diese „Nanoschichten“ in Zellen eindringen, und ihr Membrandurchtritt wurde experimentell und durch Simulationen untersucht.
Titov und Mitarbeiter führten grobkörnige Molekulardynamik-Simulationen durch, um die Interaktion von kleinen Graphen- und Few-Layer-Graphen (FLG)-Nanoschichten mit einer Lipid-Doppelschicht zu untersuchen, und berichteten über stabile Graphen-Lipid-Hybridstrukturen. Guo et al. untersuchten das Durchdringen kleiner Graphen-Nanoschichten durch Lipid-Doppelschichten und deren Auswirkungen auf die Membrandeforation.
Über die grundlegenden zellulären Interaktionen von Graphenmaterialien mit mikrometergroßen lateralen Abmessungen (Graphen-Mikroschichten), die einen Schwerpunkt in der aktuellen Entwicklung von Graphenmaterialien darstellen, ist viel weniger bekannt. Die Dringlichkeit, Richtlinien für die Herstellung sicherer biomedizinischer Diagnostika und Therapien zu entwickeln, sowie die berufliche und umweltbedingte Exposition zu regulieren, rechtfertigt erhebliche Bemühungen, die Mechanismen der Zellmembraninteraktion mit Kohlenstoffnanomaterialien, insbesondere mit Graphen-Mikroschichten, aufzuklären.
Frühere Studien zum Eindringen von Kohlenstoffnanoröhren in Zellmembranen haben darauf hingewiesen, dass es eine kritische strukturelle Dimension in der Größenordnung der Doppelschichtdicke (etwa 4 nm) gibt. Unterhalb dieser Dimension wird ein direktes Eindringen in die Doppelschicht möglich, während oberhalb dieser Schwelle eine rezeptorvermittelte Endozytose für die Aufnahme erforderlich ist. Graphen ist in einer Dimension atomar dünn, jedoch in zwei weiteren Dimensionen typischerweise groß. Unsere ersten Modellierungsarbeiten zeigten hohe Energiebarrieren für das Eindringen in die Membran, selbst wenn Monolagen-Schichten kante-zuerst auf die Lipiddoppelschicht treffen.
Diese Eintrittsbarrieren erwiesen sich als noch größer für Mehrschicht-Graphenmaterialien, die aufgrund ihrer großflächigen Herstellung durch Exfoliation von besonderem Interesse für unsere Studie sind. In der Anfangsphase war unklar, welche Arten von Graphenmaterialien (mit welcher Schichtanzahl und welchen lateralen Abmessungen) in der Lage wären, in Zellen einzudringen, und durch welchen biophysikalischen Mechanismus dies erfolgen könnte.
Daher führten wir eine kombinierte experimentelle und rechnergestützte Studie zu Graphen-Membran-Interaktionen durch, bei der wir Molekulardynamik-Simulationen und In-vitro-Zellbildgebung einsetzten, mit einem Schwerpunkt auf der Geometrie und Dynamik des Eindringens und der Aufnahme in Zellen. Die Simulationen verwendeten ähnliche grobkörnige Methoden wie in unserer früheren Studie zum Spitzen-Eindringen von Kohlenstoffnanoröhren und verwandten 1D-Materialien (30), erforderten jedoch aufgrund der atomaren Dicke von Graphen eine höhere räumliche Auflösung.
Wir untersuchen die atomaren Mechanismen der Zellinteraktion mit Few-Layer-Graphen-Mikroschichten mithilfe von grobkörnigen Molekulardynamik-Simulationen (CGMD) und all-atom gesteuerten Molekulardynamik-Simulationen (SMD). Die CGMD-Simulationen zeigen den dynamischen Prozess der Graphen-Doppelschicht-Interaktion, während die SMD-Simulationen es ermöglichen, die Energiebarrieren für das initiale Eindringen von Graphen zu bestimmen. SMD-Simulationen sagen voraus, dass idealisierte Graphenschichten mit glatten Kanten Lipiddoppelschichten bei Raumtemperatur aufgrund hoher Energiebarrieren nicht durchdringen können, selbst wenn der Erstkontakt streng kante-zuerst erfolgt.
Die Untersuchung tatsächlich hergestellter Graphen-Mikroschichten zeigt jedoch hochgradig unregelmäßige Kantentopographien, weshalb wir ein separates theoretisches Modell für die Interaktion von Graphen mit Doppelschichten entwickelten, das an scharfen Ecken oder Vorsprüngen initiiert wird. Dieses Modell sagt voraus, dass die Zellaufnahme durch ein spontanes lokales Durchstechen der Membran an Ecken oder Rauigkeiten eingeleitet werden kann, gefolgt von einer spontanen Ausbreitung entlang der Graphenkante, um eine vollständige Penetration zu erreichen.
Diese neuen Modellvorhersagen wurden durch konfokale Fluoreszenz-Bioimaging und Elektronenmikroskopie validiert. Diese zeigen ein Eindringen kante-zuerst, oder ecke-zuerst, in die Membran, sowie eine vollständige Internalisierung für eine Reihe von Few-Layer-Graphen-Mikroschichten und drei Zelltypen.
Ergebnisse und Diskussion – Die grobkörnige Molekulardynamik (CGMD)
Abbildung 1 zeigt die Ergebnisse der grobkörnigen Molekulardynamik-Simulationen (CGMD) zur Interaktion von Graphen und Few-Layer-Graphen (FLG) mit Lipid-Doppelschichten. Im ersten Simulationssatz wurde eine kleine, rhombische Monolagen-Graphenflocke mit einer Kantenlänge von 6,4 nm in einem Abstand von etwa 4 nm oberhalb und parallel zu einem quadratischen Lipid-Doppelschicht-Patch positioniert, der aus 992 Lipidmolekülen und 67.817 Wassermolekülen in einer kubischen Box mit einer Kantenlänge von 24 nm besteht (Abb. 1A). Periodische Randbedingungen wurden in allen drei Dimensionen angewandt.
Unter thermischen Fluktuationen zeigt die Graphenflocke Brownsche Bewegung, einschließlich schneller Vibrationen, Rotation und Migration in der Nähe der Doppelschicht. Ein spontanes Eindringen in die Doppelschicht wurde beobachtet, sobald die Flocke eine Konfiguration erreichte, bei der eine ihrer schärfsten Ecken nahezu orthogonal zur Membran ausgerichtet war (Abb. 1B und C). Dieses Eindringen wird durch die attraktiven Wechselwirkungen zwischen Graphen und den Schwanzgruppen der Lipide erleichtert, tritt jedoch nur auf, wenn die Spitze der durchdringenden Ecke den hydrophoben Kern der Doppelschicht berührt.
In unseren CG-Simulationen konnte die Lipidmembran vollständig von der Graphenflocke durchdrungen werden, begleitet von einer Neigung der Flocke, um die maximale Abdeckung mit dem Membraninneren zu erreichen. Aufgrund ihrer kleinen Dimensionen blieb die Graphenflocke in unserer Simulation schließlich in der Doppelschicht eingebettet.
Eine Simulation einer rhombischen Graphenflocke mit zwei unterschiedlichen Eckwinkeln zeigte, dass das orthogonale Eindringen der schärfsten Ecke die niedrigste Energiebarriere aufweist und der bevorzugte Eintrittsweg ist. Die Kantenebenen von Graphen besitzen eine komplexe Chemie und sind typischerweise mit hydrophilen Sauerstoffgruppen funktionalisiert. In Zellkulturmedien können außerdem adsorbierte Proteine auf Graphen vorliegen, die die scheinbare Hydrophobizität reduzieren können. Die GFN-Proben, die in unseren Bioimaging-Studien verwendet wurden, hatten C/O-Verhältnisse von 10 bis 32 gemäß Röntgen-Photoelektronenspektroskopie, wobei die Sauerstoffatome vermutlich an Kanten- und Defektstellen gebunden sind. Zusätzlich bedeckte die Menge an adsorbierten Proteinen je nach Bedingungen und Probe 3–46 % der GFN-Oberfläche.
Um diese Effekte zu untersuchen, wurden weitere CGMD-Simulationen mit Graphenflocken unterschiedlicher Form und Oberflächenchemie (unterschiedliche Ecken-/Kantenfunktionalisierungen sowie hydrophile/hydrophobe Eigenschaften) durchgeführt. Die Ergebnisse bestätigen, dass kleine Graphenschichten dazu neigen, über ein spontanes Eindringen an ihrer schärfsten hydrophoben Ecke in die Zelle zu gelangen. Eine Reihe von Berechnungen mit unterschiedlichen Wechselwirkungsparametern zwischen Graphen- und Lipidmolekülen zeigte außerdem, dass der Eintrittsmodus über die Ecke in einem breiten Bereich dissipativer Partikeldynamik-Parameter robust ist.
Unsere Analyse zeigt, dass die nahezu orthogonale Ausrichtung einer scharfen Graphenecke in Bezug auf die Doppelschicht die freie Wechselwirkungsenergie minimiert und thermodynamisch die bevorzugte Konfiguration darstellt, noch bevor das Eindringen beginnt.

(A–H) zeigen grobkörnige Molekulardynamik-Simulationen der Interaktionen zwischen einer Lipid-Doppelschicht und (A–D) einer kleinen Graphenflocke sowie (E–H) einer großen fünfschichtigen Graphenplatte mit gestaffeltem Stapeln und aufgerauter Kantentopografie. (A–D): Zeitsequenzen, die die Dynamik der Interaktion einer kleinen Graphenflocke mit der Lipid-Doppelschicht zeigen. Die Graphenflocke beginnt mit thermischer Bewegung, richtet sich aus, und ein scharfer Eckpunkt durchdringt schließlich die Membran, woraufhin die Flocke vollständig eingebettet wird.(E): Experimentelle Darstellung einer Graphenkantenstruktur, die durch früher dokumentierte Studien (34–36) charakterisiert wurde und als Referenz für die Simulationen dient.(F–H): Zeitsequenzen für eine große Graphenplatte mit mehreren Schichten.
Die Simulation verdeutlicht, dass raue und gestaffelte Kanten eine bevorzugte Interaktionsstelle mit der Lipidmembran darstellen. Ein scharfer Vorsprung beginnt den Penetrationsprozess, der schließlich zur Internalisierung führt. (I) zeigt die normalisierte freie Energie des Systems als Funktion der Graphenausrichtung. Hierbei wurde einer der schärfsten Eckpunkte in einer festen Distanz von 0,5 nm über der Doppelschicht positioniert. Die Energielandschaft demonstriert, dass die Ausrichtung des Graphens mit einer nahezu orthogonalen Orientierung zur Membran energetisch bevorzugt ist, was das Eindringen erleichtert. Diese Ergebnisse unterstreichen die entscheidende Rolle von Kanten- und Eckentopografien bei der Interaktion von Graphenmaterialien mit Lipidmembranen und geben Einblicke in die physikalisch-chemischen Mechanismen, die die Penetration steuern.
Wir führten auch CGMD-Simulationen von großen Graphenblättern mit wenigen Schichten durch, die mit Lipiddoppelschichten wechselwirken (Abb. 1 F-H). Zunächst untersuchten wir eine ideale, atomar glatte, unendliche Graphenkante, die mit Lipiddoppelschichten wechselwirkt, konnten aber keine Penetration beobachten.
Wir stellten fest, dass die Kanten von experimentell hergestelltem Graphen eine Rauheit auf atomarer Ebene aufweisen (Abb. 1E), wie sie in der atomar aufgelösten Rastertunnelmikroskopie zu sehen ist, und die meisten Graphenblätter mit nur wenigen Schichten weisen ebenfalls sehr raue Kanten auf (siehe Abb. 4), sowie terrassenförmige oder abgeschrägte Kantenstrukturen, die zu einer der beiden Seiten hin immer dünner werden. Abb. 1F zeigt ein Modell einer terrassierten Kantenstruktur, die an einer fünfschichtigen FLG-Flocke in Wechselwirkung mit einer Doppelschicht erzeugt wurde.
Das Simulationssystem besteht aus einem Fleck einer Doppelschicht mit 2.016 Lipidmolekülen und 133.052 Wassermolekülen in einem kubischen Kasten mit den Abmessungen 24 nm × 48 nm × 24 nm. Das plattenförmige FLG, das eine Topographie mit gezackten Kanten aufweist, die die in Experimenten beobachteten Topographien nachahmt, besteht aus fünf Atomschichten mit einem Gleichgewichtsabstand von 0,34 nm zwischen den Schichten, die anfänglich in einem Abstand von etwa 3 nm oberhalb und orthogonal zur Doppelschicht angeordnet sind. Jeder Graphenschicht ist eine andere Farbe zugeordnet. Die ersten beiden und die letzten beiden Schichten sind symmetrisch zur Mittelschicht angeordnet.
Um einen Teil einer viel größeren Struktur zu imitieren, für die die Brownsche Bewegung begrenzt ist, wird der obere Rand des FLG eingespannt und es werden in allen drei Dimensionen des Simulationskastens periodische Randbedingungen festgelegt. Die Lipiddoppelschicht macht in der Nähe des großen Graphenrandes 2,19 μs lang eine Brownsche Bewegung durch, die durch ein harmonisches Potential begrenzt wird. Letzteres wird dann entfernt, und die Doppelschichtmembran wird zur Wechselwirkung mit der zerklüfteten Graphenkante freigesetzt. Es zeigt sich, dass das FLG trotz seiner Größe in die Doppelschicht eindringt, wobei es zunächst an scharfen Ausstülpungen entlang der Kante lokalisiert eindringt. Der eingedrungene Teil der Membran breitet sich dann entlang der gesamten Kante aus, was zu einer vollständigen Durchdringung führt.
Bei diesem Prozess wird die Energiebarriere für das Eindringen durch lokales Einstechen an scharfen Ecken entlang der nominell flachen Kante überwunden, und das vollständige Eindringen wird durch die attraktive Wechselwirkung zwischen dem Graphen und den Schwanzgruppen der Lipide vorangetrieben, sobald das anfängliche Einstechen erfolgreich ist. Wir haben die Robustheit dieses Eintrittsmodus getestet, indem wir weitere CGMD-Simulationen an ein- oder mehrschichtigen Graphenflocken mit einem isolierten Vorsprung oder einer Terrasse durchgeführt haben, oder, indem wir den Kontakt in der Nähe einer Ecke oder einer lokal gefalteten Kante initiierten.
Die Simulationsergebnisse zeigen alle ähnliche Wege, die durch lokales Einstechen an einem atomar dünnen Graphenmerkmal eingeleitet werden, was nur thermische Energie zur Überwindung der kleinen Barriere erfordert, gefolgt von Ausbreitung und vollständiger Penetration, angetrieben durch hydrophobe Kräfte zwischen dem Graphen und dem Doppelschichtkern. Wir glauben, dass dieser Eintrittsmechanismus für die Zellaufnahme aller hydrophoben 2D-Nanomaterialien mit atomarer Dicke allgemein gültig sein könnte.
Die Bedeutung des lokalen Durchstoßens von Ecken für die Einleitung des Eintritts kann auch durch die Durchführung von Simulationen an idealen, atomar glatten, unendlichen Graphenkanten ohne die in realen Proben beobachteten unregelmäßigen Merkmale nachgewiesen werden. In diesem Fall zeigen unsere Simulationen, dass die Lipiddoppelschicht aufgrund einer Kombination aus einer starken Eintrittsenergiebarriere und entropischen Wechselwirkungen zwischen der Doppelschicht und einer atomar glatten Graphenkante von der Graphenkante abgestoßen wird.
Diese letzte Simulation wurde durchgeführt, um die Bedeutung der lokalen Durchdringung als auslösendes Ereignis weiter zu demonstrieren, entspricht aber keinem bekannten biologischen Expositionsszenarium, da sie einen Zellkontakt mit einer einheitlichen, atomar glatten, horizontal ausgerichteten, langen Graphenkantenstruktur erfordern würde, die in der Praxis nur schwer zu erreichen ist.
Unsere Simulationen zeigen ein lokales orthogonales Durchstechen an einer scharfen Ecke oder Unebenheit, das das Eindringen von Graphen in Lipiddoppelschichten einleitet. Um dieses Verhalten besser zu verstehen, verwenden wir die Technik der thermodynamischen Integration, um die freie Energie des Systems als Funktion von zwei Orientierungswinkeln einer rhombischen Graphenflocke zu berechnen, wenn eine der scharfen Ecken der Flocke in einem Abstand von 0,4 nm über der Doppelschicht fixiert ist. Dabei ist der Winkel zwischen der langen Diagonalachse der Flocke und der Doppelschicht innerhalb der Graphenebene und der Winkel zwischen den Vektoren normal zur Graphenebene und der Membranebene (Abb. 1I). Die mit dem orthogonalen Durchstechen der Ecken verbundene freie Energie wird als Referenzwert auf 0 gesetzt.
Es überrascht nicht, dass die Orientierung, die der Graphenflocke parallel zur Doppelschichtebene entspricht, die höchste freie Energie aufweist, da sie die stärkste Begrenzung der thermischen Bewegung in dieser Konfiguration bewirkt. Wir haben die berechnete freie Energie mit ihrem Spitzenwert in der parallelen Konfiguration normalisiert. Die aufgetragene normalisierte freie Energie in Abb. 1I zeigt, dass die orthogonale Ausrichtung die niedrigste freie Energie aufweist, da sie die thermischen Bewegungen von Membran und Graphen am wenigsten einschränkt (und damit die Entropie des Systems maximiert).
Simulationen für alle Atome
Unsere CGMD-Simulationen deuten darauf hin, dass lokalisiertes Eckendurchstechen eine entscheidende Rolle während der Anfangsphase der Zellaufnahme von Graphen spielt. Um die Energieentwicklung zu bestimmen, die mit solchen anfänglichen Durchdringungsereignissen verbunden ist, werden weitere Simulationen an einem All-Atom-Modell der Eckendurchdringung einer einlagigen Graphen-Flocke durch einen Doppelschicht-Patch aus 1-Palmitoyl-2-oleoyl-sn-glycero-3-phosphocholin (POPC) Lipid in einer Box mit Wassermolekülen durchgeführt. Die All-Atom-Simulationen sind in zwei Typen unterteilt. Mit den Simulationen vom Typ I soll getestet werden, ob es eine positive treibende Kraft für die Durchdringung in einer MD-Simulation mit allen Atomen gibt.
Wie in Abb. 2A gezeigt, wurde beobachtet, dass sich ein dreieckiges Graphenplättchen, das anfänglich in einer die Ecken durchdringenden Konfiguration quer durch die Doppelschicht platziert wurde, spontan nach unten bewegt und weiter in die Doppelschicht eindringt. In Typ-II-Simulationen wird die Energiebarriere, die mit dem Durchstoßen der Ecken verbunden ist, durch SMD-Simulationen berechnet, bei denen eine Graphenecke durch eine virtuelle Feder über die Doppelschicht gezogen wird. Abb. 2B zeigt die aus den SMD-Simulationen berechnete Graphen-Bilayer-Wechselwirkungsenergie als Funktion des Durchdringungsabstands.
Die SMD-Berechnungen bestätigen, dass die Energiebarriere gering ist, nur ∼5kBT, damit die Graphenecke die obere hydrophile Kopfregion der Doppelschicht durchstoßen kann. Kurz nach diesem Punkt beginnt die gesamte Wechselwirkungsenergie aufgrund günstiger Wechselwirkungen zwischen den Lipidschwänzen im Kern der Doppelschicht und einer immer größer werdenden Fläche von eingetauchtem Graphen zu sinken. Sowohl unsere CGMD- als auch unsere All-Atom-Simulationen zeigen, dass das Durchdringen der Ecken mit einer kleinen Energiebarriere verbunden ist, die mit der thermischen Energie vergleichbar ist, und im Wesentlichen ein spontaner Prozess ist.

Molekulardynamik-Simulationen des Eckendurchbruchs einer Graphen-Monolage durch eine Lipid-Doppelschicht. (A) Simulationen, die direkt zeigen, dass das Durchstoßen der Ecke spontan erfolgt. (B) Graphen-Bilayer-Wechselwirkungsenergie als Funktion der Penetrationsdistanz, die das Vorhandensein einer Energiebarriere von etwa 5kBT im Zusammenhang mit dem Durchstoßen der Ecke zeigt. Der Mittelwert der Wechselwirkungsenergie wurde aus 11 unabhängigen Simulationsläufen ermittelt, die Fehlerbalken zeigen den SD. Die relativ großen Fluktuationen der Wechselwirkungsenergie bei großer Penetrationsdistanz sind hauptsächlich auf zufällige Translations- und Rotationsbewegungen des Graphens relativ zur Doppelschichtmembran und zufällige Konfigurationsänderungen einzelner Lipide neben dem Graphen zurückzuführen. (C) Analytisches Modell des Eckendurchbruchs.
Sowohl CGMD-, als auch All-Atom-Simulationen zeigen, dass die Struktur der Lipid-Doppelschicht nach dem Einfügen von Graphen im Wesentlichen intakt bleibt, mit der Ausnahme, dass die hydrophoben Schwänze der Lipide aufgrund der starken Adhäsion an den Seitenflächen von Graphen etwas begradigt werden. In einer Kontinuumsbeschreibung kann die mit dem Durchstechen einhergehende Energieänderung durch vier Variablen ausgedrückt werden: hH und hT (Dicken der Kopf- und Schwanzgruppen in der Lipidmonoschicht, wie in Abb. 2C gezeigt) und γH und γT (Wechselwirkungs-Energiedichten zwischen einer Seitenfläche von Graphen und den Kopf- und Schwanzgruppen der Lipide im Verhältnis zu derjenigen zwischen Lösungsmittel und Graphen). Für eine Graphenecke mit einem Innenwinkel von 2α kann die Energieänderungsrate beim Durchstechen als stückweise Funktion der Eindringtiefe h wie folgt beschrieben werden.

Unsere in Abb. 2A gezeigte All-Atom-Simulation deutet darauf hin, dass es eine positive treibende Kraft für das letzte Regime des Durchstoßens gibt. Unter dieser Bedingung ist zu erkennen, dass die Energie im ersten Regime ansteigt, während sie in beiden Regimen und abnimmt. Die Energiespitze tritt auf, wenn die Graphen-Spitze im hydrophoben Kern liegt, an einem Punkt, der durch definiert ist. Daraus ergibt sich die Energiebarriere für das Durchstechen bei einer kritischen Eindringtiefe von (…) Wenn man (…) und (…) nimmt, zeigt sich, dass die Energiebarriere für das Durchstechen der Ecken bei einer kritischen Eindringtiefe von liegt. Diese Zahlen stimmen ausgezeichnet mit den in Abb. 2B gezeigten Ergebnissen der direkten SMD-Simulation überein.
Es mag verlockend sein, eine Analogie zwischen dem hier beschriebenen Einstechen von Graphenecken in eine Lipiddoppelschicht und dem mechanischen Einstechen durch Spannungskonzentration an scharfen Ecken herzustellen. Wir betonen jedoch, dass es sich bei ersterem um einen im Wesentlichen spontanen Prozess handelt, während letzterer durch eine angewandte Kraft angetrieben wird.
Experimente zur Biobildgebung
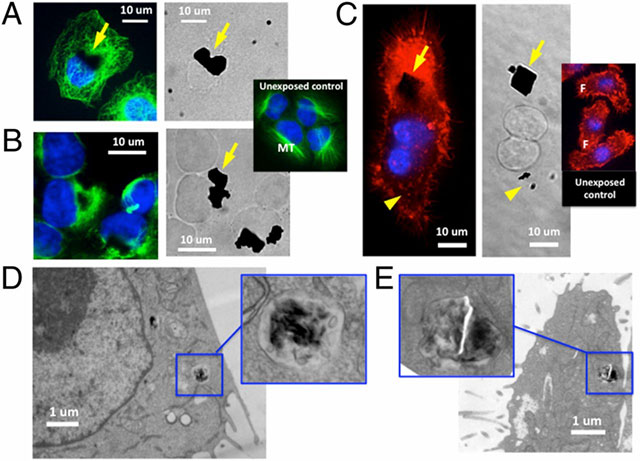
Abb. 3 und 4 zeigen konfokale Fluoreszenz- und Ex-situ-Elektronenmikroskopaufnahmen, die die MD-Vorhersagen der ersten Durchdringung von Kanten/Ecken und des Eindringens von Zellen in Graphen-Mikroschichten bestätigen. Lungenepithelzellen und Keratinozyten sind repräsentativ für die epitheliale Auskleidung der Atemwege bzw. der Haut und bilden in vitro flache, einzellige Monolagen (Abb. 3). Polarisierte Epithelzellen haben ein gut organisiertes mikrotubuläres Zytoskelett-Netzwerk, während Makrophagen eine subkortikale Verteilung von Aktinfilamenten aufweisen, die mit indirekter Immunfluoreszenz-Konfokalmikroskopie sichtbar gemacht werden kann (Abb. 3 A-C, Einschübe). Plattenförmige Graphen-Mikroschichten werden von menschlichen Lungenepithelzellen (Abb. 3 A und B) und Makrophagen (Abb. 3C) internalisiert und können mit konfokaler Bildgebung im Zytoplasma sichtbar gemacht werden. Interessant ist, dass die basalen Graphenebenen eine bevorzugte Ausrichtung parallel zur basolateralen, mit dem Substrat verbundenen Zelloberfläche aufweisen.
Plattenförmige Graphen-Mikroschichten stören die zytoskelettale Organisation sowohl von Lungenepithelzellen (Abb. 3 A und B) als auch von Makrophagen (Abb. 3C) physisch. In Dünnschnitten mit der Transmissionselektronenmikroskopie (TEM) können einige Graphen-Mikroblätter in zytoplasmatischen Vakuolen innerhalb von Makrophagen (Abb. 3D) und Lungenepithelzellen (Abb. 3E) sichtbar gemacht werden. Die Gesamtstruktur und die Integrität der subzellulären Organellen bleiben erhalten, wie TEM zeigt (Abb. 3 D und E), was unsere In-vitro-Tests bestätigt, die eine erhaltene Lebensfähigkeit der Zellen zeigen (SI Text und Abb. S8).

Zelluläre Aufnahme und Internalisierung von Graphen-Mikroblättern mit wenigen Schichten. (A-C) Konfokale Bilder von menschlichen Lungenepithelzellen (A und B) und Mausmakrophagen (C), die Graphen-Mikroschichten (0,5- bis 25-μm laterale Abmessung) nach 24 h bzw. 5 h ausgesetzt wurden. Die Zellkerne in A und B sind mit 4′,6-Diamidino-2-Phenylindol (DAPI) sichtbar gemacht (blaue Fluoreszenz). Die Mikrotubuli der Lungenepithelzellen (A und B) werden mit Antitubulin-beta-Antikörpern, die mit FITC konjugiert sind, sichtbar gemacht (grüne Fluoreszenz), während das Aktin-Zytoskelett der Makrophagen in C mit Rhodamin-Phalloidin (rote Fluoreszenz) sichtbar gemacht wird.
In nicht exponierten Lungenepithelzellen (A und B, Inset) bilden zytoplasmatische Mikrotubuli (MT) ein lineares Netzwerk, das das Zytoplasma überspannt. Internalisierte Graphenflocken (gelbe Pfeile, A und B) verdrängen das lineare Mikrotubuli-Netzwerk physisch. In nicht exponierten Makrophagen (C, Inset) ist filamentöses Aktin (F) in Aggregaten unter der Plasmamembran organisiert. Internalisierte Graphenflocken mit großer lateraler Abmessung (gelber Pfeil, C) induzieren dichte Aggregate von Aktinfilamenten, während Graphenblätter im Submikronbereich (gelbe Pfeilspitze, C) das Aktinzytoskelett nicht stören. Transmissionselektronenmikroskopische Aufnahmen von Makrophagen (D) und Lungenepithelzellen (E), die 5 und 24 Stunden lang 10 ppm FLG-Folien (∼800 nm in lateraler Dimension) ausgesetzt waren, zeigen die Lokalisierung im Zytoplasma innerhalb von membrangebundenen Vakuolen (blaue Einsätze). Graphen-Mikroschichten in Vakuolen erscheinen als elektronendichte lineare Abschnitte (D, Inset) oder unregelmäßige Flocken (E, Inset).

Wechselwirkungen zwischen Zellmembranen und Graphen-Mikroschichten, die für jeden der drei Zelltypen die Durchdringung von Kanten oder Ecken zeigen. (A) Eckpenetration eines Graphenblatts von mikrometergroßer lateraler Abmessung auf der Oberfläche einer menschlichen Lungenepithelzelle bei niedriger und hoher Vergrößerung. (B) Kantenpenetration mehrerer Mikroblätter (G) in einen Makrophagen (M). (C) Kantenpenetration eines 5-μm-Blattes, das mit primären menschlichen Keratinozyten interagiert, wobei der Kanteneintritt an einer Unebenheit oder einem Vorsprung (dicker gelber Pfeil) entstanden zu sein scheint. (D) Eckendurchdringungsmodus an der Oberfläche einer primären menschlichen Keratinozyte.
Die geneigte, aufrechte Ausrichtung des Graphenblatts erzeugt in einigen Bildern subtile E-Strahl-Schatten unmittelbar neben dem Blatt. Eine sehr unregelmäßige Randtopographie ist auf fast allen Graphenplatten zu sehen. Alle Bilder sind rasterelektronenmikroskopische Feldemissionsaufnahmen von fixierten Zellen mit Osmiumtetroxid-Nachfixierung. Die Belichtungszeit beträgt 24 Stunden, mit Ausnahme der Makrophagen, die 5 Stunden belichtet wurden. Die Zellen in A und B wurden während der Probenvorbereitung für die Rasterelektronenmikroskopie nicht der kritischen Punkttrocknung unterzogen. Die Graphen-Mikroschichten hier und in Abb. 3 weisen Schichtzahlen von 4 bis 25 auf (Skalenbalken, 2 μm).
Die in Abb. 3 verwendeten Bildgebungsprotokolle sind nützlich, um die Graphen-Internalisierung und -Orientierung zu zeigen, geben aber keinen Aufschluss über den Eintrittsmodus. Daher führten wir kürzere Zeitbelichtungen durch, um den Aufnahmeprozess zu erfassen, indem wir Ex-situ-Feldemissions-SEM von Zielzellen verwendeten, deren äußere Membran durch Osmiumtetroxid-Nachfixierung verstärkt wurde. Diese Aufnahmen wurden sowohl mit, als auch ohne, Trocknung am kritischen Punkt durchgeführt, um Trocknungsartefakte zu vermeiden. Abb. 4 zeigt hochauflösende Bilder von Zelloberflächen, die nach 5 bzw. 24 Stunden Graphen ausgesetzt wurden. In allen Fällen und für alle drei Zielzelltypen ist eine Durchdringung der Membran am Rand oder in den Ecken zu erkennen. Abb. 4 C und D zeigen besonders deutliche Fälle von Membranpenetration, die an einer Unebenheit oder einem Vorsprung am Graphenrand (Abb. 4C) oder an einer Graphenecke (Abb. 4D) begonnen zu haben scheint.
Man beachte, dass die Ränder dieser Blätter sehr unregelmäßig sind (Abb. 4 A, C und D) und zahlreiche Stellen für die anfängliche Penetration bieten, wie in der Modellierung beschrieben . Die Internalisierung dieser plättchenförmigen Graphen-Mikroschichten mit einer lateralen Abmessung von 0,5 nm bis zu 5 μm (Tabelle S1) beeinträchtigte die Lebensfähigkeit der Zellen bei den in diesen In-vitro-Studien verwendeten Dosen und Zeitpunkten nicht. Generell können jedoch inhalierte Nanopartikel mit gesundheitsschädlichen Wirkungen in Verbindung gebracht werden (41), und Schinwald et al. berichteten, dass Graphen-Nanoplättchen bei Mäusen nach einer Rachensaugung die Bildung von Granulomen und eine Lungenentzündung auslösten.
Die intrazelluläre Aufnahme und zytoplasmatische Lokalisierung von plättchenförmigen Graphen-Nanomaterialien kann die Organisation des Zytoskeletts (Abb. 3 A-C) und normale physiologische Funktionen wie polarisierte Sekretion, Barrierebildung und Zellmigration während der Differenzierung und Reparatur von Epithelverletzungen beeinträchtigen. Es hat sich gezeigt, dass die Internalisierung von Nanopartikeln durch Makrophagen die Phagozytose und die Beseitigung von Partikeln und Mikroben aus der Lunge stört. Schinwald et al. weisen nach, dass Graphen-Nanoplättchen nicht ohne Weiteres aus der Lunge entfernt werden und die Freisetzung von proinflammatorischen Mediatoren aus Makrophagen induzieren. Andere Studien berichten, dass Graphen-Nanomaterialien biokompatibel sind. Es sind noch viele weitere Arbeiten erforderlich, bevor die Gesundheitsrisiken von Graphen-Materialien vollständig bewertet werden können.
Zusammenfassend wurden grobkörnige Molekulardynamik, atomgesteuerte MD, analytische Modellierung sowie Live-Zell- und Ex-situ-Bioimaging eingesetzt, um die grundlegenden Mechanismen der Interaktion von Graphen mit Lipiddoppelschichten zu untersuchen. Die Simulationen zeigen ein direktes Eindringen in die Doppelschicht, das mit einer lokalen Durchdringung an scharfen Ecken oder an Vorsprüngen entlang der Graphenkanten beginnt, gefolgt von einer Ausbreitung entlang der Kante, um eine vollständige Durchdringung zu erreichen. Bei einem kleinen Graphenplättchen führen Brownsche Bewegung und entropische Antriebskräfte im membrannahen Bereich dazu, dass das Plättchen zunächst orthogonal zur Ebene der Doppelschicht positioniert wird, was dann zu spontanem Durchstoßen der Ecken führt.
Mit Hilfe von Molekulardynamiksimulationen mit allen Atomen wird die Entwicklung der freien Energie beim Durchdringen der Ecken verfolgt, und es zeigt sich nur eine kleine Energiebarriere, vergleichbar mit kBT. Interessanterweise hat die Zellmembran in Abwesenheit von scharfen Ecken oder Kantenvorsprüngen eine hohe intrinsische Energiebarriere gegen das Durchdringen von langen Graphen-Kantensegmenten, obwohl diese atomar dünn sind. Solche gleichmäßigen, atomar glatten, horizontal ausgerichteten, langen Graphenkanten sind jedoch selten, so dass in der Praxis die Zellpenetration spontan erfolgt, da die Kantenrauhigkeit im Atom- oder Nanomaßstab vorhanden ist und die Energiebarriere im Wesentlichen beseitigt.
Experimentelle Bildgebungsstudien bestätigen das Eindringen von Graphen in Zellmembranen in einem dominanten „edge-first“- oder „corner-first“-Modus für jede der drei untersuchten Zellarten: Lungenepithelzellen, Keratinozyten und Makrophagen. Die Experimente zeigen auch das Eindringen und die erfolgreiche Aufnahme von GFN-Flocken mit einer Größe von 5-10 μm in der lateralen Dimension, was die Modellvorhersage unterstützt, dass die Barrieren für die Aktivierung des Eindringens aufgrund der Initiierung an lokalen scharfen Merkmalen nicht intrinsisch längenabhängig sind.
Mit Hilfe von Molekulardynamiksimulationen mit allen Atomen wird die Entwicklung der freien Energie beim Durchdringen der Ecken verfolgt, und es zeigt sich nur eine kleine Energiebarriere, vergleichbar mit kBT. Interessanterweise hat die Zellmembran in Abwesenheit von scharfen Ecken oder Kantenvorsprüngen eine hohe intrinsische Energiebarriere gegen das Durchdringen von langen Graphen-Kantensegmenten, obwohl diese atomar dünn sind. Solche gleichmäßigen, atomar glatten, horizontal ausgerichteten, langen Graphenkanten sind jedoch selten, so dass in der Praxis die Zellpenetration spontan erfolgt, da die Kantenrauhigkeit im Atom- oder Nanomaßstab vorhanden ist und die Energiebarriere im Wesentlichen beseitigt. Experimentelle Bildgebungsstudien bestätigen das Eindringen von Graphen in Zellmembranen in einem dominanten „edge-first“- oder „corner-first“-Modus für jede der drei untersuchten Zellarten: Lungenepithelzellen, Keratinozyten und Makrophagen.
Die Experimente zeigen auch das Eindringen und die erfolgreiche Aufnahme von GFN-Flocken mit einer Größe von 5-10 μm in der lateralen Dimension, was die Modellvorhersage unterstützt, dass die Aktivierungsbarrieren für das Eindringen aufgrund der Initiierung an lokalen scharfen Merkmalen nicht intrinsisch längenabhängig sind. Sobald die anfängliche Energiebarriere für die spontane Membranpenetration überwunden ist, stellen wir die Hypothese auf, dass die Wechselwirkung zwischen den hydrophoben Basalflächen der Graphen-Mikroblätter und dem inneren hydrophoben Bereich der Plasmamembran die zelluläre Aufnahme fördert. Hydrophobe Oberflächen werden als schädigungsassoziierte molekulare Muster (DAMPs) angesehen, die unspezifisch die angeborene Immunantwort aktivieren.
Hydrophobe zelluläre Oberflächen und oberflächenfunktionalisierte Nanopartikel werden leichter internalisiert und lösen eine stärkere angeborene Immunantwort aus als schwach geladene, hydrophile Oberflächen. Aufgrund dieses Mechanismus stellen wir die Hypothese auf, dass Graphen-Mikroblätter, die in hydrophobe Lipiddomänen eindringen, von Zielzellen als DAMPs erkannt werden können, die die erste Verteidigungslinie gegen Partikel und Mikroben darstellen, die sich nach dem Einatmen auf der Haut oder der epithelialen Auskleidung der Lunge absetzen. Die hier sowohl experimentell als auch durch Simulationen dokumentierte Fähigkeit von Graphen-Mikroblättern mit großer lateraler Ausdehnung, in Zellen einzudringen, könnte zu einer Störung des Zytoskeletts, einer Beeinträchtigung der Zellmotilität, einer Beeinträchtigung der epithelialen Barrierefunktion, oder anderen geometrischen und sterischen Effekten führen, die weitere Untersuchungen benötigen.
Materialien und Methoden – Die grobkörnige MD-Simulationen
Die grobkörnigen Simulationen in dieser Arbeit basieren auf DPD, einer Lagrange-Methode, die von der Grobkörnigkeit der Molekulardynamik abgeleitet ist und als mesoskopische Simulationsmethode für Biomembransysteme weit verbreitet ist. Die Lipid-Doppelschichtmembran wird durch das grobkörnige Modell H3(T5)2 dargestellt. Die hydrophilen Lipidköpfe und hydrophoben Lipidschwänze sind als rote, bzw. gelbe, Kügelchen dargestellt. Eine Einheitszelle von CG-Graphen besteht aus drei Kügelchen mit einem Abstand zwischen den nächsten Nachbarn von 0,4 nm und einem Innenwinkel von 60°. Die Wechselwirkungen zwischen den Kügelchen sind so gewählt, dass die elastischen Eigenschaften von CG-Graphen mit den experimentellen Ergebnissen übereinstimmen. Die Wechselwirkungsparameter zwischen Graphen und Lipidmolekülen werden anhand von Parametern aus All-Atom-MD-Simulationen kalibriert und auch in einem Bereich variiert, um die Robustheit der wichtigsten Beobachtungen zu testen. Die Simulationen werden in Anzahl-Volumen-Temperatur-Ensembles (NVT) mit dem Zeitschritt as durchgeführt und mit dem Softwarepaket LAMMPS durchgeführt.
All-Atom MD Simulationen
Die allatomaren MD-Simulationen wurden in NAMD durchgeführt und in VMD visualisiert. Das Graphen und die POPC-Lipid-Doppelschichtmembran wurden mit dem VMD Graphene Builder bzw. Membrane Builder erzeugt. Das Graphen- und Lipid-Doppelschichtsystem wurde dann mit VMD vollständig hydriert (Hinzufügen von Wassermolekülen). Das CHARMM36-Kraftfeld mit zusätzlichen Parametern für das Graphen- und TIP3-Wassermodell wurde für die Simulationen verwendet. Die Simulationen bestanden aus zwei Gleichgewichtsschritten und einem Produktionsschritt. Da die mit dem Membran-Plug-in von VMD erzeugten Lipidmembranen weit vom Gleichgewichtszustand entfernt waren, wurde ein Gleichgewichtslauf von 0,5 ns durchgeführt, bei dem alles außer den Lipidschwänzen fixiert wurde.
Dieser erste Schritt ermöglichte es den Lipidschwänzen, zu einer flüssigkeitsähnlichen Konfiguration zu verschmelzen. Im zweiten Schritt wurden die Systeme etwa 2 ns lang im Anzahl-Druck-Temperatur-Ensemble (NPT) bei einer Temperatur von 310 K und einem Druck von 1 atm ins Gleichgewicht gebracht. Als dritter Schritt wurden die Produktionsläufe durchgeführt. In allen drei Simulationsschritten wurden die Freiheitsgrade der Kohlenstoffatome der Graphenflocken außerhalb der Ebene durch harmonische Beschränkungen eingeschränkt.
Experimente zur Biobildgebung
Die Experimente wurden mit drei Zelltypen durchgeführt: Mäuse-Makrophagen, Lungenepithelzellen und primäre menschliche Keratinozyten. Die Zellen wurden kommerziellen Graphen-Mikroschichtproben mit unterschiedlichen lateralen Abmessungen (0,5-10 μm) und Schichtzahlen ausgesetzt. Die FLG-Proben wurden mit 1,2-Dipalmitoyl-sn-glycero-3-phosphocholin (DPPC) (Avanti Polar lipids)/Albumin dispergiert, bevor sie in das Zellkulturmedium gegeben wurden (Abb. S10). Einhundert Mikroliter Graphenflocken in einer 5-mg/ml-Ethanol-Stammlösung wurden zu 62 μl einer 40-mg/ml-DPPC/Ethanol-Lösung gegeben und 5 s lang geschüttelt, gefolgt von der Zugabe von 838 μl PBS mit 2 % (Vol./Vol.) Albumin, um eine 500 μg/ml (ppm) Graphen-Stammlösung mit 2,5 mg DPPC und 2 % Albumin/PBS zu erhalten.
Die Menge des adsorbierten Proteins, die sich aus der Anwendung dieses Protokolls ergibt, wurde in separaten Kontrollexperimenten gemessen und betrug je nach Bedingungen und Probe 3-46 % der GFN-Oberfläche (SI-Text). Für Keratinozyten wurde die Graphen-Stammlösung auf 10 μg/mL Graphen in Dermalzell-Basalmedium verdünnt, das Keratinozyten-Wachstumssupplemente enthält, während RPMI 1640 als Verdünnungsmedium für Makrophagen und Lungenepithelzellen diente.
Konfokale Fluoreszenzmikroskopie
Lungenepithelzellen wurden 24 Stunden lang Graphenflocken ausgesetzt. Die Zellen wurden gewaschen und mit einem Antitubulin-Antikörper gefärbt, der mit Fluorescein-Isothiocyanat (FITC) (Cell Signaling; 3623S) und 4′,6-Diamidino-2-Phenylindol (DAPI) konjugiert war. Die Makrophagen wurden wie oben beschrieben 5 Stunden lang mit Graphen in Kontakt gebracht. Die Zellen wurden gewaschen und mit Rhodamin-konjugiertem Phalloidin und DAPI gefärbt. Die Bilder wurden mit einem Olympus konfokalen Fluoreszenzmikroskop mit Drehscheibe (Modell IX81) und motorisiertem Umkehrmikroskop visualisiert, um die Aufnahme und die Organisation des Zytoskeletts zu beurteilen.
Elektronenmikroskopie
Für die TEM-Probenvorbereitung wurden zwei verschiedene Methoden verwendet, um zu überprüfen, ob die Graphenflocken von den Zellen aufgenommen werden. Die Zellen wurden in Karnovskys Fixiermittel [5 % (Vol./Vol.) Glutaraldehyd, 4 % (Vol./Vol.) Formaldehyd in 0,1 M Natriumcacodylat-Puffer, pH 7,4] (Electron Microscopy Sciences; 11650) bei 4 °C und anschließend dreimal mit 0,1 M Natriumcacodylat-Puffer gespült und in 2 % (Vol./Vol.) wässrigem Osmiumtetroxid (Electron Microscopy Sciences; RT 19152) nachfixiert. Die Proben wurden in eiskalten, abgestuften Ethanollösungen und anschließend in eiskaltem, wasserfreiem Aceton dehydriert. Die Infiltration der Proben erfolgte mit Durcupan ACM-Harz (Electron Microscopy Sciences) mit abnehmenden Anteilen von Aceton und schließlich mit Durcupan allein.
Elektronenmikroskopie
Für die TEM-Probenvorbereitung wurden zwei verschiedene Methoden verwendet, um zu überprüfen, ob die Graphenflocken von den Zellen aufgenommen werden. Die Zellen wurden in Karnovskys Fixiermittel [5 % (Vol./Vol.) Glutaraldehyd, 4 % (Vol./Vol.) Formaldehyd in 0,1 M Natriumcacodylat-Puffer, pH 7,4] (Electron Microscopy Sciences; 11650) bei 4 °C und anschließend dreimal mit 0,1 M Natriumcacodylat-Puffer gespült und in 2 % (Vol./Vol.) wässrigem Osmiumtetroxid (Electron Microscopy Sciences; RT 19152) nachfixiert. Die Proben wurden in eiskalten, abgestuften Ethanollösungen und anschließend in eiskaltem, wasserfreiem Aceton dehydriert. Die Infiltration der Proben erfolgte mit Durcupan ACM-Harz (Electron Microscopy Sciences) mit abnehmenden Anteilen von Aceton und schließlich mit Durcupan allein.
Die Zellen wurden in Durcupan eingebettet und 48 Stunden lang bei 60 °C polymerisiert. Die Zellblöcke wurden auf einem Reichert Ultracut Ultramikrotom entweder mit einem Diamantmesser (unbehandelte Zellen) oder einem Glasmesser bei 80 nm dünn geschnitten. Die Schnitte wurden auf Kupfergitter gelegt und in einem Philips 410 Transmissionselektronenmikroskop mit einer Advantage HR CCD-Kamera betrachtet. Die Bilder wurden mit der Bildgebungssoftware „Advanced Microscopy Techniques“ aufgenommen.
Die intrazelluläre Lokalisierung von Graphen-Nanoblättern wurde auch durch Schneiden von Zellen bestätigt, die in einer Monolage auf Glasdeckgläsern gewachsen waren. Für die Rasterelektronenmikroskopie wurden die Zellen 30 Minuten lang in 2 % (Vol./Vol.) wässrigem Osmiumtetroxid postfixiert und anschließend in 25 % (Vol./Vol.), 50 % (Vol./Vol.), 70 % (Vol./Vol.), 2 × 95 % (Vol./Vol.) und 3 × 100 % (Vol./Vol.) Ethanol dehydriert. Nach dem Trocknen am kritischen Punkt gemäß den Anweisungen des Herstellers (Ladd Research) wurden die Deckgläser mit Gold gesputtert und mit einem Feldemissions-SEM (LEO 1530-VP) untersucht.
Quelle: PNAS
Quellen: PublicDomain/legitim.ch am 05.12.2024




















Der Körper wurde gekapert und die DNA und DNS überschrieben.
Es sind keine vom Schöpfer erschaffenen Menschen mehr, sondern androide Wesen, welche über keine silberne Schnur mehr verfügen werden.
Eine Kaperung des Ganzen, verhindert auch sämtliche Verbindungen. Egal ob geistig oder sonst welche.
Es wird durch die Überschreitung alles abgeschaltet, was zur Erkenntnis der Liebe führt.
Es werden zum Schluß monotone Geräte sein, ohne Empathie, welche auf jede neurolinguistische Programmierung hören werden.
Zu wahren Gefühlen nicht mehr fähig, so wie auch das Tier, the Beast, die KI keine wahren Gefühle kennt.
Sie wünschen sich zu sterben und sehnen sich nach dem Tod. Doch der Tod wird vor ihnen fliehen.
Denn der Tod ist quasi eine Erlösung aus dem Zustand von Materie, Jedoch nicht bei künstlich digitalisierten Geräten, welche eines natürlichen Geschöpfes nicht mehr zuzuordnen sind, da es keine Verbindung mehr gibt.
So wie es einer der Kommentierenden Mario Salmhofer vortrefflich auf den Punkt gebracht hat.
👇
https://www.pravda-tv.com/2024/12/geist-ueber-materie-wie-unser-bewusstsein-die-materielle-realitaet-erschafft-video/#comment-361947
Der Tot ist der Schutzgeist der menschlichen Rasse!
🖕
menschlich.!!!
Für Geräte und Sachen ist der Schutzgeist nicht zuständig